Введение. Инфекции мочевыводящих путей (ИМП), пиелонефриты, циститы и уретриты, относятся к числу широко распространенных бактериальных инфекций человека [1]. Они существенно снижают качество жизни пациента и трудно поддаются лечению. Эти инфекции особенно актуальны для женщин: по статистике, около половины женского населения имели хотя бы один эпизод урологической инфекции в течение жизни [1]. Возникновение неосложненных ИМП чаще всего связано с восходящей инфекцией энтеропатогенов, преодолевающих естественный защитный барьер мочеполовой системы, который в основном обеспечивается нормофлорой, в первую очередь лактобациллами влагалища [2–4]. Уменьшение количества лактобацилл в нормофлоре человека служит фактором риска развития ИМП [5]. Лечение неосложненной ИМП проще других инфекций, поскольку в подавляющем большинстве случаев можно установить возбудителя, причем чаще всего заболевание вызвано моноинфекцией; при осложненной ИМП очень часто имеются причины, поддерживающие инфекционный процесс: обструкция, конкременты и др. [1].
К основным возбудителям ИМП относятся уропатогенные Escherichia coli (УПЕК), которые характеризуются высокой генетической и фенотипической гетерогенностью [6]. Кроме того, они способны образовывать биопленки, обеспечивающие их защиту от иммунной системы макроорганизма и антимикробных препаратов [7].
Цель данной работы: охарактеризовать микробиологический и молекулярно-генетический профили резистентности штаммов E. coli.
Материалы и методы
Биоэтические требования
Материалы, использованные в данном исследовании, не содержат персональных данных пациентов: в описании клинических изолятов отсутствуют фамилии и имена пациентов, их адреса и номера историй болезней. В то же время в соответствии с требованиями Биоэтического комитета Российской Федерации каждый пациент подписывал информированное согласие при поступлении в лечебное учреждение на проведение лабораторных исследований.
Штаммы
Штаммы E. coli (n=18) были выделены в рамках пилотного одноцентрового исследования из мочи женщин в возрасте 23–84 лет, проходивших лечение в урологическом отделении Инфекционной клинической больницы № 1 Ярославля в декабре 2016 – январе 2017 г. Для видовой идентификации бактерий осуществляли посевы на дифференциально-диагностические питательные среды Агар Эндо-ГРМ, агар Клиглера-ГРМ, железо-глюкозо-лактозный агар с мочевиной и ацетатный агар (ФБУН ГНЦ ПМБ, Оболенск, Россия) согласно инструкции производителя с последующим подтверждением видовой принадлежности бактерий на приборе MALDI-TOF Biotyper («Bruker», Германия).
Питательные среды, условия культивирования и хранения
Культуры штаммов E. coli выращивали на плотной питательной среде ГРМ 1 и жидкой питательной среде ГРМБ (ФБУН ГНЦ ПМБ, Оболенск, Россия) в аэробных условиях при температуре 37°C. Хранили культуры при при температуре -70°C в 20%-ном растворе глицерина.
Оценка подвижности бактерий
Подвижность E. coli определяли по методике [8] с изменениями. Готовили бактериальную взвесь (108 КОЕ/мл) в среде ГРМБ, культивировали 4 ч при температуре 37°С с аэрацией (100 об/мин), разводили физиологическим раствором (ФР) в 1000 раз, 10 мкл наносили в центр чашки Петри с 0,3%-ным агаром на основе ГРМБ, культивировали 24 ч при температуре 37°С, не переворачивая чашки Петри. Подвижность бактерий оценивали, измеряя диаметр зоны распространения бактерий в толще полужидкого агара в мм. Эксперимент проводили трижды. В качестве отрицательного контроля использовали штамм E. coli С600, полученный из Государственной коллекции патогенных микроорганизмов и клеточных культур «ГКПМ-Оболенск».
Определение колициногенности
Продукцию колицинов клетками E. coli выявляли методом двухслойного агара [9]. Наличие колициногенности у штамма определяли как наличие зоны задержки роста тест-штамма шириной 2 мм и более. В качестве тест-штамма, чувствительного к колицинам, также использовали штамм E. coli С600.
Определение чувствительности к антагонистическому действию лактобактерий
Для этого использовали штамм Lactobacillus acidophilus, выделенный из пробиотического препарата Лактобактерин (НПО «Биомед», Россия), рекомендованного в качестве интравагинального применения. Антагонистическую активность штамма исследовали методом агаровых слоев. Штамм считали чувствительным к антагонистическому действию лактобактерий при наличии зоны задержки роста шириной 1 мм и более [4].
Биопленкообразование
Способность штаммов к образованию биопленок оценивали по методике [10] с изменениями: 30 мл жидкой питательной среды ГРМБ инокулировали одной полной бактериологической петлей (1 мкл) ночной агаровой культуры, вносили в среду 3 полипропиленовых купона (3 см2), инкубировали при температуре 37°С с аэрацией 150 об/мин на шейкере Unimax 1010 («Heidolph Instruments», Германия) в течение 24 ч. После этого купоны дважды отмывали в ФР и обрабатывали ультразвуком на гомогенизаторе Soniprep 150 («MSE Ltd», Великобритания) при амплитуде 2×10-6 м в течение 2 мин. Полученную суспензию клеток титровали и делали высевы на плотную питательную среду. Плотность биопленки рассчитывали как количество КОЕ на единицу площади (см2). В качестве штаммов сравнения использовали референс-штамм ATCC25922 и лабораторный штамм E. coli C600.
Чувствительность к антибактериальным препаратам
Минимальные подавляющие концентрации (МПК) 17 антибактериальных препаратов 6 функциональных классов: β-лактамов – ампициллина (ОАО «Синтез», РФ), ампициллина/сульбактама (ОАО «Красфарма», РФ), амоксициллина/клавуланата («Sandoz», Германия), цефуроксима («GlaxoSmithKline», Италия), цефотаксима («Aventis Pharma Ltd.», Великобритания), цефтазидима (ОАО «Синтез», РФ), азтреонама («Sanofi», Франция), меропенема (ОАО «Красфарма», РФ); фторхинолонов – ципрофлоксацина (ОАО «Синтез», РФ), левофлоксацина (ОАО «Красфарма», РФ), норфлоксацина (ЗАО «Фармацевтическое предприятие "Оболенское"», РФ); аминогликозидов – гентамицина (РУП «Белмедпрепарат», Республика Беларусь); фосфомицина («Zambon Switzerland Ltd.», Швейцария); нитрофуранов – фуразолидона (ОАО «Борисовский завод медицинских препаратов», РБ), фурацилина (ООО «Авексима Сибирь», РФ), нитрофурантоина (ОАО «Ирбитский химфармзавод», РФ) и полимиксинов – колистина (ООО «НВЦ Агроветзащита С.-П.», РФ) определяли методом микроразведений в бульоне [11]. Результаты интерпретировали в соответствии с рекомендациями Европейского комитета по определению чувствительности к антимикробным препаратам EUCAST Breakpoint tables v 9.0 (http://www.eucast.org). В качестве стандартного использовали штамм E. coli ATCC25922, полученный из Государственной коллекции патогенных микроорганизмов и клеточных культур «ГКПМ-Оболенск».
Чувствительность к дезинфектантам
Минимальные бактерицидные концентрации (МБК) 10 дезинфектантов: перекись водорода (ООО «Инновация», РФ), надуксусная кислота (ООО «НПО СпецСинтез», РФ), этанол (ЗАО «ЭКОлаб», РФ), пропанол-1 (OOO «АО РЕАХИМ», РФ), пропанол-2 («AppliChem GmbH», Германия), хлоргексидин («Sigma-Aldrich», США), N,N-бис (3-аминопропил)-додециламин (ООО «Химреактив М», РФ), бензалкониум хлорид («Sigma-Aldrich», США), триклозан («Sigma-Aldrich», США), глутаровый альдегид (OOO «АО РЕАХИМ», РФ), определяли методом микроразведений в бульоне с последующим высевом на плотную питательную среду [12]. Пороговую концентрацию (cut-off) для каждого дезинфектанта определяли как наибольшее значение МБК, зафиксированное для исследуемых штаммов E. coli [13].
Детекция генов антибиотикорезистентности
Методом ПЦР со специфичными праймерами определяли гены β-лактамаз blaTEM-, blaSHV-, blaCTX-M-, blaOXA-48- и blaNDM-типов, а также интегроны классов 1 и 2 [14–18].
Секвенирование последовательностей генов
Последовательности ДНК секвенировали в ООО «СИНТОЛ» (Москва) и анализировали с помощью программ Vector NTI 9 («Life Technologies», США), Chromas (Technelysium, http://technelysium.com.au/wp/chromas/) и BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi).
Полногеномное секвенирование
Полногеномное секвенирование штаммов E. coli проводили на платформе Illumina MiSeq с использованием наборов Nextera DNA Library Preparation Kit («Illumina», Карлсбад, США) и MiSeq Reagent Kits v3 («Illumina», Карлсбад, США), согласно инструкции производителя. Полученные единичные прочтения собирали в контиги, используя программное обеспечение SPAdes 3.9.0 [19]. Собранные геномы аннотировали с помощью сервера NCBI Prokaryotic Genome Annotation Pipeline [20]. В работе использовали инструменты Центра геномной эпидемиологии (Center for Genomic Epidemiology, http://www.genomicepidemiology.org/): поиск нуклеотидных последовательностей генов устойчивости к антибактериальным препаратам проводили с помощью сервиса ResFinder 3.1 [21]. Полногеномные последовательности размещены в базе данных GenBank (NZ_SERW00000000.1, NZ_SERS00000000.1, NZ_SERU00000000.1, NZ_SERV00000000.1, NZ_SERX00000000.1, NZ_SERY00000000.1, NZ_SERT00000000.1).
Статистический анализ
Статистическую обработку экспериментальных данных проводили с помощью стандартных функций пакета Microsoft Office, 2010. Различие сформированных групп подтверждено методом двухвыборочного t-критерия Стьюдента для независимых выборок при коэффициенте достоверности p<0,01.
Результаты
Анализ клинических данных
Пилотное одноцентровое исследование проводили в лечебном учреждении, которое исследует ежемесячно 400 образцов мочи. В период исследования с 13.12.2016 по 09.01.2017 было выделено 87 культур бактерий, в том числе Enterococcus faecalis (n=26), Escherichia coli (n=18), Klebsiella pneumoniae (n=18), Proteus mirabilis (n=6), Streptococcus agalactiae (n=3) и других (n=16). Штаммы E. coli были получены от пациенток женского пола в возрасте 23–84 лет с диагнозами «хронический цистит» (n=11), «цистит» (n=2), «инфекция мочевыводящей системы» (n=3), «мочекаменная болезнь» (n=1) и «гиперактивный мочевой пузырь» (n=1). Сведения о применении антибиотикотерапии до взятия проб отсутствовали.
Подвижность, колициногенность, биопленкообразование и чувствительность к антагонистическому действию Lactobacillus acidophilus
Показатель степени подвижности клеток изучаемых штаммов E. coli (диаметр зоны распространения бактерий в полужидком агаре) существенно различался для ряда штаммов: для трех штаммов он составил 11–13 мм, для семи – 19–45 мм, для четырех штаммов – 60–74 мм; у пяти штаммов зона распространения отсутствовала – клетки этих штаммов не обладали подвижностью (табл. 1, см. рисунок, а).

Колициногенность отдельных штаммов E. coli также различалась: пять штаммов были охарактеризованы как продуценты колицинов, к которым был чувствителен тест-штамм E. coli C600. При этом зоны задержки роста тест-штамма имели ширину от 2 до 8 мм. Остальные штаммы колициногенности в отношении штамма E coli C600 не проявляли (см. табл. 1, см. рисунок, б).
Все изученные штаммы E. coli формировали биопленки на полипропиленовых купонах. Плотность клеток в биопленках после выращивания в течение 24 ч существенно различалась: 14 штаммов формировали биопленки низкой плотности (1×106±0,7×106 КОЕ/см2), два штамма – средней плотности (6×106±0,06×106 КОЕ/см2), два штамма – высокой плотности (2×107±0,2×107 КОЕ/см2) (табл. 1). Различие сформированных групп подтверждено методом двухвыборочного t-критерия Стьюдента для независимых выборок при коэффициенте достоверности p<0,01. Штаммы сравнения E. coli ATCC25922 и E. coli C600 формировали биопленки с плотностью 3×106 и 5×106 КОЕ/см2 соответственно, то есть со средней плотностью.
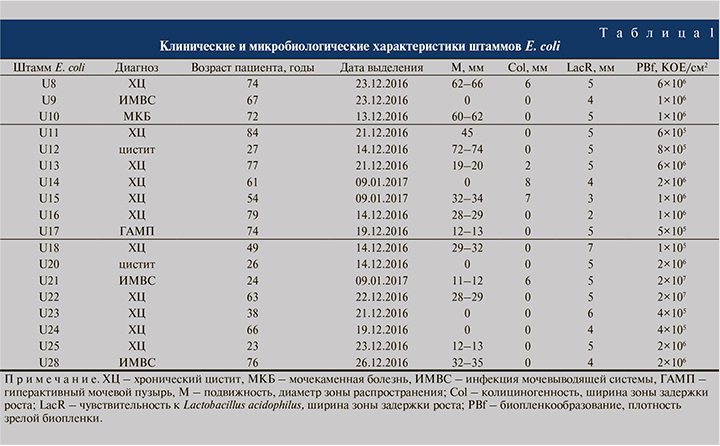
Показано, что все изученные штаммы E. coli были чувствительными к антагонистическому действию штамма Lactobacillus acidophilus. Оно выражалось в наличии зоны задержки роста тест-культуры вокруг колонии, образованной лактобактериями (см. рисунок, в). Ширина зон задержки роста варьировалась у разных штаммов от 2 до 7 мм при измерении от края колонии (см. табл. 1).
Чувствительность к антимикробным препаратам
Показано, что изучаемые штаммы E. coli были гетерогенны по чувствительности к антибактериальным препаратам (АП): семь штаммов отнесены к категории множественно лекарственно устойчивых (МЛУ), резистентных к трем и более функциональным классам АП, в том числе по два штамма к четырем и пяти классам АП. Все изученные штаммы были устойчивыми к двум нитрофуранам (фуразолидону и фурацилину) и чувствительными к третьему нитрофурану – нитрофурантоину. Большинство штаммов (n=16) были устойчивыми к β-лактамам (кроме меропенема); 7 штаммов – к фторхинолонам; 4 – к аминогликозидам; 2 штамма – к колистину. Все штаммы были чувствительными к фосфомицину (табл. 2).
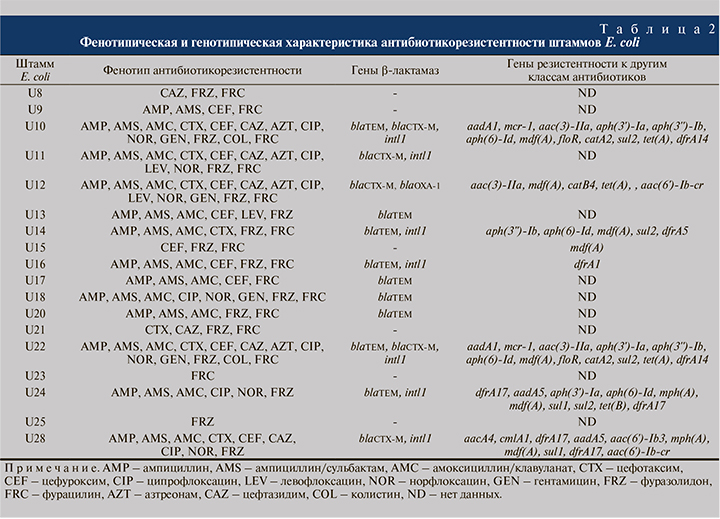
Генетические детерминанты антибиотикорезистентности
Методом ПЦР в 9 исследованных штаммах идентифицированы гены blaTEM, в 5 штаммах гены blaCTX-M; гены blaSHV, blaKPC, blaOXA-48 и blaNDM не обнаружены. Обращает на себя внимание тот факт, что гены устойчивости к β-лактамам присутствуют в большинстве изученных штаммов (n=12), причем в трех штаммах были выявлены одновременно гены двух типов β-лактамаз (blaCTX-M+blaTEM) и (blaCTX-M+blaOXA-1) (табл. 2). В семи штаммах выявлены интегроны класса 1, три из которых несли наборы генных кассет устойчивости к аминогликозидам, сульфаниламидам и хлорамфениколу; интегроны класса 2 не обнаружены. В геномах штаммов, исследованных с помощью полногеномного секвенирования, дополнительно было выявлено большое разнообразие генетических детерминант устойчивости: к β-лактамам (ген β-лактамазы blaOXA-1), аминогликозидам (гены аминогликозидацетилтрансфераз aac, аминогликозидфосфотрансфераз aph и аминогликозидаденилилтрансфераз aad), макролидам (гены макролидфосфотрансфераз mph и МЛУ эффлюксного насоса mdf), фениколам (гены МЛУ эффлюксных насосов floR и cmlA, хлорамфеникол-о-ацетилтрансферазы catB, катехол-1,2-диоксигеназы catA), сульфаниламидам (ген дигидроптероат-синтазы sul), тетрациклинам (оперон тетрациклинового эффлюксного насоса tet), триметоприму (ген дигидрофолатредуктазы dfrA) и фторхинолонам (ген фторхинолон-ацетил аминогликозид-6’-N-ацетилтрансферазы aac(6’)-Ib-cr). В двух штаммах выявлен ген устойчивости к колистину mcr-1 (см. табл. 2).
Чувствительность к дезинфектантам
Показано, что все изучаемые штаммы E. coli были чувствительными к представителям основных функциональных классов дезинфектантов (окислители, спирты, гуанидины, амины, четвертичные аммониевые соединения, альдегиды и фенолы) со следующими значениями cut-off: 0,05% к перекиси водорода, 4,4% к этанолу, 3,1% к пропанолу-1, 12,5% к пропанолу-2, 0,03% к хлоргексидину, 0,002% к амину, 0,04% к бензалкониуму хлориду, 0,25% к глутаровому альдегиду, 0,02% к надуксусной кислоте, 0,0004% к триклозану (табл. 3).

Обсуждение
E. coli – один из ведущих возбудителей урологических инфекций, выделенных в рамках пилотного одноцентрового исследования от пациентов женского пола в возрасте от 23 до 84 лет. Среди 18 охарактеризованных штаммов E coli была отмечена значительная гетерогенность по фенотипическим признакам и наборам детектированных генетических детерминант. Важным фенотипическим признаком для УПЕК является подвижность, поскольку это свойство связано со способностью распространяться по мочевыводящим путям и обеспечивать феномен восходящей инфекции. Более половины охарактеризованных нами штаммов имели высокий показатель подвижности – диаметр зоны их распространения в полужидком агаре составил 19–74 мм, что существенно превысило аналогичный показатель для референсных УПЕК штаммов E. coli CFT073 (13–14 мм) и J96 (10–11 мм) в опубликованном ранее исследовании X. Yang et al. [8]. Другой важный фенотипический признак УПЕК – колициногенность, способность бактерий продуцировать бактериоцины, угнетающие рост других штаммов E. coli, что обеспечивает им селективные преимущества. Доля колициногенных штаммов в нашем исследовании составила ~30%, что сопоставимо с долей колициногенных штаммов в популяциях УПЕК (10–25%) [22, 23].
Еще одним важным для УПЕК свойством является способность формировать биопленки, поскольку бактерии в составе биопленок более устойчивы к лекарственным препаратам и к воздействию защитных механизмов макроорганизма [1]. Все изученные в нашей работе штаммы формировали на купонах из промышленного полистирола биопленки различного уровня плотности – от 105 до 107 КОЕ/см2, что аналогично данным, полученным для бактериальных культур E. coli Ampr, выделенных от пациентов с инфекциями мочевыводящих путей [24]. К важным свойствам УПЕК относится также способность противостоять антагонистическому действию лактобактерий, которые составляют значительную часть нормофлоры влагалища и дистального отдела уретры [2, 3]. В нашем исследовании отмечена довольно низкая чувствительность исследуемых штаммов к антагонистическому действию лактобацилл (ширина зон задержки роста составила 2–7 мм), что несколько меньше представленного в работе эстонских авторов показателя (6–10 мм) [4].
Анализ чувствительности штаммов E. coli к клинически значимым АП показал, что более трети штаммов относились к категории МЛУ, что сопоставимо с долей МЛУ штаммов, зафиксированной в международном исследовании Northern Dimension Antibiotic Resistance Study (NoDARS) для Российской Федерации, – 26,9% [25]. Все штаммы в нашем исследовании были чувствительными только к нитрофурантоину, фосфомицину и меропенему, что незначительно отличается от данных исследования NoDARS, в котором доля штаммов, чувствительных к нитрофурантоину и фосфомицину, составляла по 99%, а к меропенему – 100% [25]. У большинства изученных в нашем исследовании штаммов идентифицированы гены β-лактамаз типов CTX-M, TEM и OXA; у половины штаммов были выявлены последовательности интегронов. Несмотря на то что тетрациклин и хлорамфеникол в настоящее время не имеют клинической значимости как препараты лечения урологических заболеваний, выявление генетических детерминант устойчивости к этим лекарствам у штаммов УПЕК важно для оценки процессов генетического обмена между бактериями, персистирующими в организме человека.
Особое внимание обращают на себя два штамма E. coli, которые характеризовались устойчивостью к резервному антибиотику колистину и несли ген mcr-1. Ранее mcr-1-позитивные штаммы УПЕК были описаны в Китае [26].
В России этот ген был недавно детектирован в штаммах E. coli в ФГБНУ «Российский научный центр хирургии им. академика Б.В. Петровского» в составе плазмиды pMCR-1_Msc (GenBank MK172815).
Важным свойством патогенных бактерий является их чувствительность к дезинфицирующим веществам, поскольку от этого зависит их способность выживать в условиях окружающей среды. Показано, что изученные в данной работе штаммы УПЕК были чувствительными к основным функциональным классам дезинфектантов и антисептиков, применяемых в лечебных учреждениях. Большинство дезсредств, размещенных на сайте Дез.ру (http://dezr.ru) на дату 05.04.2019, рекомендованных для дезинфекции и стерилизации изделий медицинского назначения, содержащих в качестве действующего вещества перекись водорода, этиловый спирт, пропанол-1, хлоргексидин, амин или надуксусную кислоту, обеспечивали достаточную концентрацию активного компонента для инактивации клеток штаммов, изученных в нашей работе. Определенные нами значения МБК и cut-off дезинфектантов против уропатогенных E. coli были аналогичными опубликованным ранее [12, 27].
Заключение
Анализ выявленных в клинических штаммах E. coli генетических детерминант антибиотикорезистентности свидетельствует о генетическом разнообразии изученных штаммов. По комплексу выявленных у штаммов фенотипических признаков можно заключить, что они обладали рядом свойств, способствующих их персистенции в мочевыводящих путях. Полученные в работе результаты показывают, что штаммы E. coli, выделенные от пациентов с диагнозами «хронический цистит», «цистит», «мочекаменная болезнь», «инфекция мочевыводящей системы» и «гиперактивный мочевой пузырь», имеют как сходные, так и различающиеся биологические и генетические характеристики. Все 18 изученных штаммов УПЕК обладали способностью образовывать биопленки in vitro; все штаммы были чувствительными к антагонистическому действию пробиотического штамма Lactobacillus acidophilus, ко всем 9 использованным в работе дезинфектантам и антисептикам, а также к антибиотикам нитрофурантоину, меропенему и фосфомицину. Важно отметить, что один из 18 штаммов, E. coli U12, выделенный от пациентки с диагнозом «цистит», был устойчив ымодновременно к 13 использованным антибиотикам: ампициллину, ампициллину/сульбактаму, амоксициллину/клавуланату, цефотаксиму, цефуроксиму, цефтазидиму, азтреонаму, ципрофлоксацину, левофлоксацину, норфлоксацину, гентамицину, фуразолидону и фурацилину; этот штамм служил носителем β-лактамаз двух типов – blaCTX-M и blaOXA-1. Полученные в ходе исследования данные по чувствительности штаммов E. coli к клинически значимым антибактериальным препаратам и дезинфектантам могут быть использованы клиницистами при выборе оптимальной антибиотикотерапии и схемы обработки абиотических поверхностей в урологических отделениях.



